STEREOFORME
Dai dati termodinamici e quelli diffrattometrici è possibile ottenere un diagramma di Ramachandran del potenziale.
È possibile una sola stereoforma, che si ottiene per valori equivalenti di rotazione della coppia: a elica (rapporto 1:1).
Il passo dell’elica (la distanza interatomica, ottenuta per diffrattometria) consiste di spirali molto più ampie dell’α-elica. Proiettandolo sull’asse dell’elica, si ha un periodo completo su una distnaza che va dai 34 Ǻ (per il DNA) a quasi il doppio (per l’RNA) con 10 nucleotidi: la struttura è molto più ampia e meno compatta dell’α-elica.
DISPOSIZIONE SPAZIALE
Nell’organizzazione spaziale delle basi azotate rispetto al monosaccaride è privilegiato l’assetto a minimo ingombro sterico (anti) con l’anello furanosico e l’anello dell’eterociclo orientati da parti opposte, rispetto a quello a massimo ingombro (sin) che crea massima repulsione tra atomi e strutture, quindi delle distorsioni.
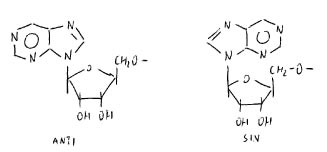
Il monosaccaride può essere il riboso o il desossiriboso.
L’OH in 2’ del riboso si trova molto vicino al legame fosfodiestereo: si realizzano quindi effetti di repulsione tra atomi con lo stesso valore di elettronegatività. Il polimero ribonucleotidico per ogni residuo ha un certo valore di distorsione dall’allineamento perfetto; nell’elica ciò è la sommatoria degli effetti di distorsione. L’elica diventa perciò più ampia e meno stabile.
Nel desossiriboso l’H in 2’ occupa uno spazio minimo e non c’è alcuna distorsione: le eliche sono più compatte, più stabili, e hanno una semivita più lunga.
Il polimero presenta lo scheletro degli atomi 5’C, 4’C e 3’C con legame fosfato (porzione polare carica) e il resto della parte furanosica e le basi azotate (struttura molto meno polare). Il polimero è quindi diviso in due regioni: una polare carica (fosfosaccaridica) e una meno polare (sviluppo della base).
Nell’RNA la zona polare si estende fino a comprendere il 2’C del riboso, mentre nel DNA il 2’C (centro metilenico) passa nello sviluppo della zona apolare, mantenendo la struttura di avvolgimento ad elica più compatta.
GERARCHIE STRUTTURALI
(1) Struttura primaria: sequenza (serie degli elementi inseriti) ⇒ DNA, RNA
(2) Struttura secondaria: elica ⇒ DNA, RNA
(3) Struttura supersecondaria: polieliche (il duplex; eventuale superavvolgimento) ⇒ DNA
(4) Struttura terziaria: ripiegamenti (transizione da filamentosa a globulare con formazione di superficie idrodinamica a contatto con l’ambiente) ⇒ RNA
(5) Struttura quaternaria: associazioni protomeriche (ad esempio nei ribosomi, i centri di sintesi delle proteine) ⇒ RNA
STABILIZZAZIONE DELL’ELICA
È affidata agli elementi che danno interazioni per ponte a idrogeno: le basi, che in qualunque polimero, essendo voluminose, sono sempre orientate equatorialmente (risultato ottenuto da studi cristallografici).
Attraverso la tautomeria, le basi sono in grado di dare interazioni deboli (ponte a idrogeno) la cui direzionalità (accettore o donatore) dipende dal microambiente e dalle sue caratteristiche, in grado di condizionare lo spostamento verso una forma tautomerica o le altre. Quando si affrontano due basi con gruppi che si complementano (uno donatore di un legame a idrogeno, l’altro accettore), si ha stabilizzazione dell’elica.
Nella fascia apolare le basi, a seconda dell’orientazione spaziale del polimero, hanno due possibilità di affrontarsi: impilamento e appaiamento.
Impilamento: nell’elica singola, interazione tra basi di nucleotidi che hanno distribuzione spaziale tale da farle sovrapporre l’una rispetto all’altra entro i limiti di distanza consentiti dalle interazioni deboli.
Appaiamento: è un sistema di stabilizzazione più forte e si realizza quando si forma una doppia elica (nel DNA) e la base di un’elica è disposta in modo da affrontare la base dell’altra elica.
1) PURINE MONOSOSTITUITE: 2 (1N, SOSTITUENTE IN 6C)
2) PURINE DISOSTITUITE: 3 (1N, SOSTITUENTE IN 2C, SOSTITUENTE IN 6C)
3) PIRIMIDINE: 3 (3N, SOSTITUENTE IN 4C, SOSTITUENTE IN 2C A MENO CHE SI ABBIA INTERAZIONE CON UNA PURINA MONOSOSTITUITA)
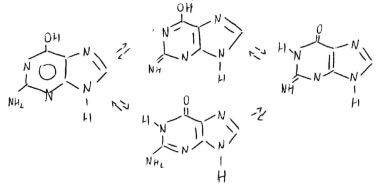
L’appaiamento è fortemente stabilizzante, ma perché avvenga il duplex dev’essere a sezione costante (la sezione cilindrica della doppia elica dev’essere costante).
UNICI APPAIAMENTI POSSIBILI (DNA)
A∙∙∙∙T (2 INTERAZIONI)
G∙∙∙∙C (3 INTERAZIONI)
Sistema di riconoscimento specifico tra basi.
UNICI APPAIAMENTI POSSIBILI (RNA)
A∙∙∙∙U (2 INTERAZIONI)
G∙∙∙∙C
Avvengono sulla catena singola, che ripiegandosi può dare tratti a duplex (anche tra zone molto lontane) transitori perché nel successivo ripiegamento la struttura viene persa. Inoltre la sezione deve sempre essere costante.
I tratti a duplex dell’RNA sono come i COIL, per la presenza di nucleotidi atipici (perché non danno interazioni a ponte idrogeno).





