Descrizione del genoma dei retrovirus
Ricoperto dalla proteina nucleocapsidica, il genoma è formato da due identiche copie di RNA. Ognuna di queste copie è costituita da una molecola RNA lineare a polarità positiva e il contenuto, l’informazione genetica, è dato da 7 fino a 10 kilobasi. Caratteristica del genoma è quella di avere all’estremità 5’ un Cap e all’estremità 3’ una coda di poli(A), quindi le due estremità hanno caratteristiche del tutto sovrapponibili a quelli che sono i messaggeri della cellula ospite.
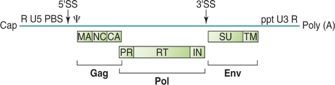
In tutti i retrovirus sono almeno presenti tre geni: il gene gag, il gene pol ed il gene env.
Env: sta per “envelope”; l’informazione di questo gene porta alla sintesi delle due proteine, che poi sono proteine glicosilate: la proteina SU con localizzazione superficiale e la proteina transmembrana TM.
Quindi tre geni indispensabili, bastano solo questi tre geni perché un retrovirus possa intraprendere un ciclo replicativo.
Continua a leggere:
- Successivo: Il ciclo replicativo dei retrovirus
- Precedente: I Retrovirus
Dettagli appunto:
-
Autore:
Simone Pisu
[Visita la sua tesi: "Caratterizzazione molecolare ed eterogenicità delle varianti emoglobiniche in Sardegna"]
- Università: Università degli Studi di Cagliari
- Facoltà: Biologia
- Corso: Biologia Cellulare e Molecolare
- Esame: Virologia molecolare
- Docente: Prof. Marongiu
Altri appunti correlati:
- Microbiologia clinica
- Biologia applicata
- Microbiologia
- Microbiologia Medica
- Filologia della letteratura italiana
Per approfondire questo argomento, consulta le Tesi:
- Piante medicinali come ausilio per contrastare le patologie virali: tre casi-studio
- Contributo allo studio ed alla terapia dell'epatite C nel paziente HIV-positivo
- Memetica e nuovi media
- Peptidi overlapping derivanti da sequenze gH-omologhe di HSV-1 come inibitori dell'attività virale.
- Selezione di Mutanti HIV-1 Resistenti e Basi Molecolari della Resistenza di Nuovi Inibitori Non-Nucleosidici della Trascrittasi Inversa
Puoi scaricare gratuitamente questo appunto in versione integrale.


