Il ciclo replicativo dei retrovirus
Eventi iniziali del ciclo replicativo
Come vanno le cose una volta che il virus si è ancorato alla cellula? Deve riconoscere dei recettori. L'antirecettore è localizzato nella proteina SU. Entra per fusione, quindi l'envelope si fonde con la membrana della cellula ospite, il core virale viene ad essere liberato nel citoplasma, si spoglia parzialmente, e a scattare subito in queste prime fasi è il funzionamento della trascrittasi inversa, enzima obbligatoriamente presente nella particella virale infettante perché un ciclo replicativo dei retrovirus possa realizzarsi. Questa trascrittasi inversa copia il genoma, formato da due molecole ognuna delle quali costituita da un singolo filamento di RNA; dopo il funzionamento della trascrittasi inversa questo RNA monocatenario è convertito in DNA bicatenario, che passa attraverso la membrana nucleare, e grazie all'intervento dell'integrasi virale, sotto forma di provirus va ad integrarsi nel genoma della cellula ospite. Adesso il virus si è spogliato di tutti i suoi rivestimenti, la sua informazione genetica da una molecola di RNA a singolo filamento è stata convertita in DNA bicatenario che si è integrato nella marea dei geni cellulari.
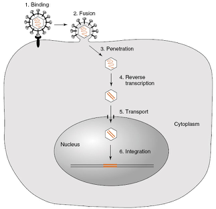
Esaminiamo questi eventi iniziali e concentriamoci nel processo di trascrittasi inversa che ci permette di vedere come si forma il provirus. Cosa deve fare la trascrittasi inversa per copiare l'RNA monocatenario in DNA bicatenario che l'integrasi integrerà nel cromosoma della cellula ospite? Deve sintetizzare dapprima una catena di DNA a singolo filamento utilizzando come stampo l'RNA genomico, quindi si forma un ibrido RNA-DNA, deve poi rimuovere l'RNA template e per ultima cosa deve sintetizzare la catena complementare di DNA per arrivare ad un DNA bicatenario. Questo ci dice che stiamo esaminando un'attività enzimatica che assolve tre differenti funzioni.
Cosa dovrà fare per sintetizzare la catena (-) di DNA sullo stampo del genoma? Dovrà comportarsi come DNA polimerasi RNA dipendente. Una volta che si forma l'ibrido deve rimuovere l'RNA genomico, fa questo grazie alla seconda attività enzimatica che è quella di RNasi H, associata alla trascrittasi inversa, che riesce a degradare il template. H sta per “ibrido” perché l'RNasi H attacca l'RNA quando è presente in forma di ibrido RNA-DNA. Degradando l'RNA rimane una molecola di DNA monocatenario, che deve essere copiata per arrivare a DNA bicatenario, ed ecco la terza funzione assolta dalla trascrittasi inversa, DNA polimerasi DNA dipendente. Quindi DNA polimerasi RNA dipendente, RNasi H, DNA polimerasi DNA dipendente.
Trascrizione inversa
Vediamo maggiormente nei dettagli molecolari come vanno le cose. Partiamo dalla parte in alto a sinistra della figura: quello che vediamo è il genoma di un retrovirus, abbiamo partendo dall'estremità 5' una sequenza R, identica e ripetuta all'estremità 3', segue U5, il PBS che lega il tRNA, i tre geni gag, pol ed env, la ppt, la U3 verso la terminazione 3' ed infine la sequenza R. Per poter funzionare la trascrittasi inversa necessita di un innesco, rappresentato da un tRNA che si lega alla sequenza PBS, quindi il tratto che vi sto indicando è un tratto di RNA a doppia catena, lo utilizza come primer e copia la sequenza U5 ed R con conseguente sintesi di U5' e R'. Questo nuovo tratto neo sintetizzato è costituito da un ibrido DNA (R'-U5') ed RNA (del genoma). Scatta la funzionalità dell'RNasi H associata alla trascrittasi inversa che digerisce questo tratto di RNA, quindi digerisce R e U5, mentre essendo presente in una doppia catena di RNA viene risparmiato il tratto PBS. A questo punto si assiste a quello che viene chiamato il “primo salto” della trascrittasi inversa: il corto segmento di DNA neosintetizzato (si tratta di DNA negativo perché si è formato sulla catena di RNA a polarità positiva) rimanendo legato al primer rappresentato dal tRNA, salta all'estremità opposta. R del genoma è presente sia ad un'estremità, sia ripetuto all'estremità opposta, ed ecco che l'R' del DNA neo sintetizzato, per complementarietà potrà legarsi all'R presente all'estremità 3' del genoma. Si è creato un primer che ha la possibilità di copiare in tutta la sua lunghezza l'RNA genomico. Siamo arrivati alla formazione di un ibrido RNA-DNA, e ancora una volta l'RNA può essere digerito dall'RNasi H associata alla trascrittasi inversa. Questa digestione dell'RNA genomico è quasi completa ad eccezione del tratto ppt che resiste alla digestione. Quindi siamo arrivati ad aver copiato una catena di DNA e la catena negativa di DNA che porta ancora legato il ppt. Qual è il ruolo del ppt? Ancora una volta la trascrittasi inversa necessita di un innesco, si mantiene questo corto frammento resistente alla digestione da parte dell'RNasi H e lo utilizza come innesco per iniziare la sintesi (funziona in questo caso l'attività enzimatica come DNA polimerasi DNA dipendente) di questo corto frammento di DNA della catena + copierà la sequenza U3', R' e U5'. Arriva a copiare fino alla fine e il risultato di questo funzionamento è la sintesi di questo corto frammento DNA+ che contiene U3, R, U5 e questo ricordate che era PBS. Arrivati a questo punto si ha quello che è chiamato il “secondo salto”. La sequenza PBS neo sintetizzata si porta all'estremità 5' dove si lega per complementarietà alla sequenza PBS'. Ancora una volta con questo salto si ha la possibilità in entrambe le catene di un innesco che ne consente il completamento di entrambe le catene. Siamo partiti da un RNA a polarità positiva e siamo arrivati ad un DNA bicatenario.
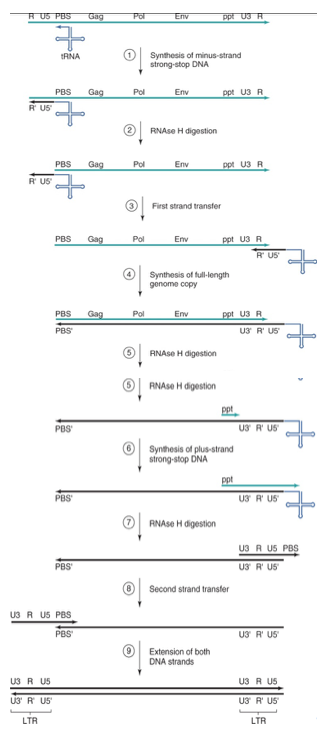

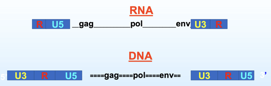
Cosa c'è di diverso tra questo DNA bicatenario che si integrerà nel cromosoma dell'ospite e l'originario genoma costituito da RNA? Il risultato è che il provirus formato dal funzionamento della trascrittasi inversa è più lungo del genoma. Molto schematicamente è riassunto in questa figura. Le estremità caratteristiche del genoma contengono una sequenza R e la sequenza unica U5 ad un'estremità, e U3 e R all'estremità opposta. A seguito dei salti che la trascrittasi inversa ha compiuto per realizzare la sintesi di quello che sarà il provirus, due sequenze identiche, U3, R e U5, vengono ad essere formate e localizzate all'estremità 5' e all'estremità 3'. Queste sequenze sono note come Long Terminal Repeats (LTR), e vedremo che svolgono un ruolo determinante nel controllare il processo di trascrizione che porterà alla sintesi, partendo dal provirus, degli RNA messaggeri virali. Quindi il provirus è più lungo, due sequenze LTR identiche sono presenti all'estremità 3' e all'estremità 5'. È importante sottolineare che la trascrittasi inversa, come le RNA polimerasi RNA dipendente che abbiamo fino ad oggi esaminato per es. nel virus dell'influenza, nel virus del morbillo, nel virus del raffreddore, è un enzima estremamente inaccurato ed è stato calcolato che, una volta che copia il genoma ad RNA, può introdurre in questo genoma ben da 1 a 10 errori, quindi un tasso mutazionale elevato, che comporta un enorme variabilità. Anche per i retrovirus, l'abbiamo esaminato anche parlando dell'HCV, la popolazione di retrovirus che è presente in un organismo non è una popolazione omogenea ma piuttosto è un insieme di varianti al quale viene dato il nome di quasispecie; è l'inaccuratezza del funzionamento della trascrittasi inversa che determina questa enorme variabilità, in casi estremi copia una molecola di RNA trasformandola in un DNA bicatenario con ben 10 errori, con una enorme conseguente variabilità.

Integrazione del DNA provirale nel DNA della cellula ospite
Siamo arrivati ad un DNA bicatenario, che a questo punto va nel nucleo e, grazie all’intervento dell’integrasi, altra attività enzimatica presente nel core virale, viene ad essere integrato nel cromosoma della cellula. A questo punto è chiamato provirus. 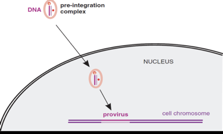

Trascrizione e traduzione
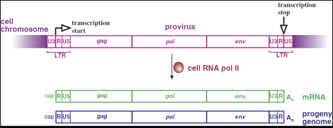
Come arriva a sintetizzare, una volta integrato, i propri mRNA? Lo fa inserendosi in quello che è il normale flusso di informazione della cellula ospite. In altre parole l'RNA polimerasi II della cellula è in grado di trascrivere i geni del provirus arrivando alla sintesi dei messaggeri cellulari. Questo che vediamo qui più scuro è il DNA della cellula e integrato è presente il provirus, il quale alle sue estremità 5' e 3' è caratterizzato dalla sequenza LTR, che contengono i 3 geni fondamentali di un retrovirus gag, pol ed env. L'RNA polimerasi della cellula sa trascrivere ed il punto di inizio della trascrizione è tra la sequenza U3 e la R, per poter trascrivere è necessaria la compartecipazione di molti fattori di trascrizione della cellula ed è proprio la disponibilità di questi fattori di trascrizione, che interagiscono con la U3, che consente ai retrovirus di esprimersi in un determinato ospite. Se la cellula ospite gli dà i fattori di trascrizione necessari, il virus potrà esprimersi, la polimerasi cellulare potrà trascrivere, arriva tranquillamente ai propri messaggeri. Se la cellula non ha a disposizione i fattori di trascrizione che si devono legare a U3, il provirus integrato non potrà esprimersi perché non potrà trascrivere i propri RNA messaggeri. Importante sottolineare che questo processo di trascrizione dà luogo sia ad mRNA che a nuovi RNA genomici. Tra U3 ed R inizia la trascrizione, mentre la poliadenilazione, quindi lo stop della trascrizione avviene all'estremità opposta tra R e U5. Questo è importante perché quando trascrive l'intera sequenza formerà degli RNA genomici identici all'originario genoma del virus, in altre parole non vengono trascritte porzioni delle LTR che servono solo come zone di controllo. Quindi arriverà alla sintesi di RNA genomici caratterizzati da un'unica sequenza U5 presente al terminale 5' ed un'unica sequenza U3 al terminale 3'.
Ecco qui il provirus, con le estremità LTR tra le quali sono presenti i tre geni gag, pol ed env. Come fa ad avere i trascritti indispensabili per la sintesi di tutte le proteine necessarie alla realizzazione del proprio ciclo replicativo? Un mezzo importante è offerto dallo splicing: tutti i retrovirus sottostanno ad almeno un processo di splicing. Come vanno le cose nei retrovirus più semplici, che hanno solo gag, pol ed env? La polimerasi cellulare copia il provirus e dà origine ad un mRNA che contiene l'informazione per la sintesi delle proteine Gag e delle proteine Pol. Questo trascritto dà origine sia ai messaggeri gag che a messaggeri gag-pol. Questo RNA, che è l'intera lunghezza dell'originario genoma virale, funge sia da messaggero per la sintesi di gag e pol, sia funge da RNA genomico. A seguito di un processo di splicing, l'espressione dei geni gag e pol viene rimossa, e ha luogo grazie a questo splicing la formazione del messaggero env, delle due proteine SU e TM, localizzate nell'envelope virale. Ricapitolando: questo è il provirus integrato, delimitato dalle LTR, la polimerasi II della cellula lo copia in tutta la sua lunghezza a partire dal punto tra U3 ed R, questi RNA a catena lunga hanno una duplice funzione: da una parte sono genomi della futura progenie virale e portano anche l'informazione che darà origine alla formazione delle proteine gag e pol. Come fa ad aumentare la propria capacità di sintetizzare più proteine? In questo caso, che è il più semplice, lo fa con un unico processo di splicing. Ha trascritto tutto il segmento del provirus, ecco che a seguito di uno splicing, si sintetizza il messaggero che gli consentirà di produrre le proteine superficiali.
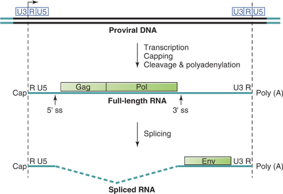

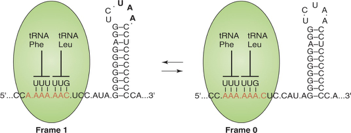
Abbiamo visto come gag e pol vengono prodotti come unico messaggero, siamo in una cellula eucariotica, in questo messaggero viene riconosciuto un unico sito di inizio a livello del promotore della sintesi proteica, e da questo precursore lungo RNA il virus ha la necessità di sintetizzare sia le proteine strutturali interne, quelle derivanti dal gene gag (la proteina matrice, la proteina nucleocapsidica e la proteina capsidica), d'altra parte deve pure, seppur in minor quantità, sintetizzare il prodotto del gene pol che porta alla sintesi della proteasi, della trascrittasi inversa e della proteasi. Un unico punto di ingresso alla sintesi proteica, come arriva a risolvere il problema di sintetizzare le proteine di cui ha bisogno? Lo fa in questa maniera. Come al solito, gli servono più proteine strutturali che non proteine ad attività enzimatica. Ecco che comincia questo lungo messaggero ad essere tradotto, e ad un certo punto nella sequenza è presente un ripiegamento a forcina. Qui vediamo l'RNA che porta l'informazione dei geni gag e pol, che ha preso contatto col ribosoma, e siamo in fase di traduzione. Ad un certo punto nell'RNA è presente una forcina, nella quale c'è questa tripletta UAA che è una tripletta di terminazione. Quando la sintesi proteica arriva a questo punto, incontra questo sito di terminazione, avremo la sintesi della proteina gag. Alcune volte però deve essere tradotta anche l'informazione genetica contenuta in pol perché un po' di attività enzimatica gli serve. Come arriva a tradurre anche l'informazione del gene pol? Quando l'mRNA scorre nei ribosomi e arriva nelle vicinanze di questo ripiegamento a forcina, ecco che il ribosoma stalla, si ferma, e nella maggior parte dei casi, dopo questo momento di stallo, riprende tranquillamente la lettura del messaggero fino a incontrare la tripletta di termine UAA dando origine al polipeptide gag; alcune volte, occasionalmente però, quando c'è questo stallo determinato dal ripiegamento a forcina, può succedere che si realizzi quello che si chiama frameshift, uno scivolamento, lo vedete qua, c'è una sequenza di 7 basi, nel nostro caso sono 6 basi A seguite da una C. Quando il ribosoma stalla sul messaggero in questo punto di lettura, fa un passo indietro e si lega non alla seconda, alla terza, alla quarta A, ma si lega alla prima, alla seconda e alla terza. Cosa succede se il ribosoma riprende il processo di traduzione da questa configurazione, che è chiamata frame 0 (zero)? Riprende la traduzione seguendo un diverso modulo di lettura. Quando le cose vanno normalmente la tripletta successiva è rappresentata da UCC. In seguito allo scivolamento, la prima tripletta che chiamerà il successivo amminoacido è CUC, è cambiato il modulo di lettura, e questo cambiamento del modulo di lettura fa sì che non venga più letto UAA come tripletta di terminazione e la sintesi proteica può procedere per arrivare alla sintesi del polipeptide gag-pol. Perché la tripletta AAA viene comunque tradotta in leucina? Perché riconosce lo stesso amminoacido; è già avvenuto il legame, nella maggior parte dei casi le cose vanno così, lega i due amminoacidi. Poi a un certo punto c'è questo ingombro, cosa può succedere? Fa un passo indietro e passiamo al frame1 (forse voleva dire 0?). Viene richiamato un altro transfer con un altro amminoacido. La tripletta è differente, cambia il modulo di lettura, non sarà più formata UAA che funge da terminatore. Perché anziché esserci UUU nell'appaiamento nella figura a destra, c'è UUG? Perché si era già legato prima. Quindi siamo arrivati a giustificare la sintesi di gag, che porta l'informazione delle proteine strutturali interne, e la sintesi di messaggeri più lunghi gag-pol che portano alla sintesi delle tre attività enzimatiche proteasi, integrasi e trascrittasi inversa.

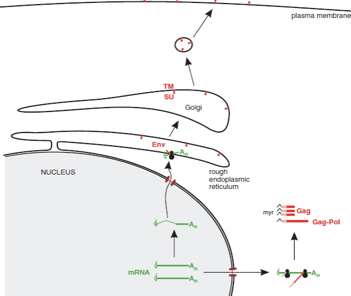
Dobbiamo ora seguire l'espressione del messaggero env che si è formato in seguito ad un processo di splicing. Mentre gag e pol sono tradotte nei ribosomi liberi nel citoplasma, ed è questo il loro prodotto che vedete qua, il messaggero env viene tradotto in ribosomi associati al reticolo endoplasmatico. Porta alla sintesi prima di un unico precursore che poi viene clivato da proteasi cellulari per dare origine alle due proteine TM e SU. Una volta che sono state sintetizzate, passando nel Golgi, queste due proteine vengono glicosilate, per poi essere posizionate sulla membrana citoplasmatica della cellula ospite (sono questi quadratini rossi in figura), e da queste porzioni nelle quali le glicoproteine virali sono state inserite, il virus potrà fuoriuscire per gemmazione. A questo punto, grazie al processo di sintesi proteica, siamo arrivati ad avere a disposizione tutte le proteine che servono al virus. Abbiamo detto che le proteine glicosilate sono inserite nella membrana citoplasmatica e che contemporaneamente nel citoplasma si è formata la proteina gag-pol e la proteina gag. Sia la proteina gag sia la proteina gag-pol alla loro estremità N-terminale vengono miristilate.

Assemblaggio
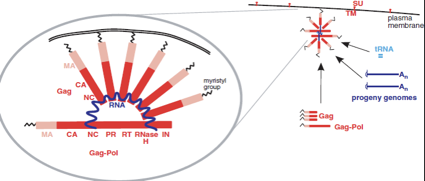
A questo punto può cominciare il processo di assemblaggio. È un processo estremamente complesso e neanche noto in tutti i dettagli. Qui abbiamo la superficie della cellula dove sono inserite TM e SU e qui abbiamo un ingrandimento di quella che è una particella virale in via di formazione. In azzurro l'RNA genomico che in forma diploide comincia a partecipare ad un complesso di assemblaggio. Le proteine gag con la loro estremità miristilata, si legano alla membrana citoplasmatica, e con l'estremità opposta (l'estremità NC) si legano ad un particolare segnale di packaging che trovano nell'RNA virale. Dapprima sono poche, poi queste proteine gag che si assemblano con l'RNA virale aumentano, contemporaneamente si sono legate anche poche copie del precursore gag-pol.
Mentre avviene la migrazione della particella neo sintetizzata verso la membrana citoplasmatica contenente TM e SU, dalla quale il virus uscirà per gemmazione, sia il precursore gag-pol che il precursore gag vengono clivati nelle proteine costituenti: gag darà origine alla proteina di matrice, alla proteina capsidica e alla nucleoproteina; pol, grazie a questo clivaggio, darà origine alle 3 proteine che troviamo nel core virale trascrittasi inversa, proteasi e integrasi. Chi è che realizza il clivaggio dei precursori gag e gag-pol? È la terza attività enzimatica, proteasi, presente in una particella di retrovirus.
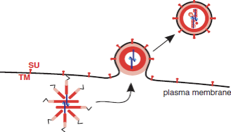

Ritorniamo un attimo indietro. In fase di sintesi proteica, grazie ad uno scivolamento si sono formate sia molte proteine gag, che poche proteine gag-pol. I precursori gag e gag-pol partecipano all'assemblaggio di una particella di un retrovirus legandosi all'RNA genomico. Quando questa particella in via di formazione sta incominciando a migrare per rivestirsi dell'envelope, rivestimento che si completerà con l'uscita per gemmazione dalla cellula, la proteasi ha il compito di tagliare questi precursori, tagliando il precursore gag-pol darà origine alle attività enzimatiche, tagliando il precursore gag darà origine alle proteine strutturali interne. Siamo arrivati alla liberazione di una particella virale ed infettante che potrà andare ad infettare una cellula vicina.
Mostra l'animazione nelle slide e la descrive ricapitolando: ecco la proteina gp120 che fino ad ora abbiamo chiamato SU, che riconosce dei recettori, un cambio di conformazione fa sì che TM esponga un potere fusogeno, ed ecco che l'envelope si fonde con la membrana citoplasmatica liberando il core; passaggio successivo è la trascrizione inversa. La trascrittasi inversa copia, si forma il DNA e l'RNasi H contemporaneamente digerisce lo stampo ad RNA. Questa catena neo formata di DNA a singolo filamento è copiata per formare DNA bicatenario, se ne va nel nucleo, passa attraverso un poro della membrana nucleare, stiamo arrivando alla formazione del provirus che si integra nel DNA nell'ospite e a questo punto il provirus può essere trascritto dall'RNA polimerasi II della cellula. Ecco qua il nostro messaggero che si sta formando. A livello del reticolo endoplasmatico avremo la formazione, la lettura del messaggero env, la proteina gag-pol invece ribosomi liberi, vengono tradotte con formazione di una poliproteina. La proteina env, ad opera di una proteasi cellulare in questo caso, nel Golgi viene clivata nelle due componenti SU e TM, che dal Golgi passeranno nella membrana citoplasmatica della cellula, per partecipare poi alla gemmazione del virus. I vari componenti strutturali si assemblano con due molecole di RNA. Intervento determinante della proteasi che cliva i precursori proteici nei vari costituenti che si assemblano, si è formata la matrice, il capside, le proteine che rivestono il genoma e siamo pronti dopo la gemmazione ad un altro ciclo replicativo.
Perché l'RNA è presente in duplice copia? Quello che vi ho mostrato durante il processo di trascrizione è uno schema semplificato, vi ho mostrato delle molecole lineari con la trascrittasi inversa che saltava da un'estremità all'altra. Molto probabilmente il processo potrebbe avvenire sotto forma circolare, e non si esclude che la trascrittasi inversa salti da un genoma all'altro.
Mostra l'animazione nelle slide e la descrive ricapitolando: ecco la proteina gp120 che fino ad ora abbiamo chiamato SU, che riconosce dei recettori, un cambio di conformazione fa sì che TM esponga un potere fusogeno, ed ecco che l'envelope si fonde con la membrana citoplasmatica liberando il core; passaggio successivo è la trascrizione inversa. La trascrittasi inversa copia, si forma il DNA e l'RNasi H contemporaneamente digerisce lo stampo ad RNA. Questa catena neo formata di DNA a singolo filamento è copiata per formare DNA bicatenario, se ne va nel nucleo, passa attraverso un poro della membrana nucleare, stiamo arrivando alla formazione del provirus che si integra nel DNA nell'ospite e a questo punto il provirus può essere trascritto dall'RNA polimerasi II della cellula. Ecco qua il nostro messaggero che si sta formando. A livello del reticolo endoplasmatico avremo la formazione, la lettura del messaggero env, la proteina gag-pol invece ribosomi liberi, vengono tradotte con formazione di una poliproteina. La proteina env, ad opera di una proteasi cellulare in questo caso, nel Golgi viene clivata nelle due componenti SU e TM, che dal Golgi passeranno nella membrana citoplasmatica della cellula, per partecipare poi alla gemmazione del virus. I vari componenti strutturali si assemblano con due molecole di RNA. Intervento determinante della proteasi che cliva i precursori proteici nei vari costituenti che si assemblano, si è formata la matrice, il capside, le proteine che rivestono il genoma e siamo pronti dopo la gemmazione ad un altro ciclo replicativo.
Perché l'RNA è presente in duplice copia? Quello che vi ho mostrato durante il processo di trascrizione è uno schema semplificato, vi ho mostrato delle molecole lineari con la trascrittasi inversa che saltava da un'estremità all'altra. Molto probabilmente il processo potrebbe avvenire sotto forma circolare, e non si esclude che la trascrittasi inversa salti da un genoma all'altro.
Continua a leggere:
- Successivo: HTLV-1
- Precedente: Descrizione del genoma dei retrovirus
Dettagli appunto:
-
Autore:
Simone Pisu
[Visita la sua tesi: "Caratterizzazione molecolare ed eterogenicità delle varianti emoglobiniche in Sardegna"]
- Università: Università degli Studi di Cagliari
- Facoltà: Biologia
- Corso: Biologia Cellulare e Molecolare
- Esame: Virologia molecolare
- Docente: Prof. Marongiu
Altri appunti correlati:
- Microbiologia clinica
- Biologia applicata
- Microbiologia
- Microbiologia Medica
- Filologia della letteratura italiana
Per approfondire questo argomento, consulta le Tesi:
- Piante medicinali come ausilio per contrastare le patologie virali: tre casi-studio
- Contributo allo studio ed alla terapia dell'epatite C nel paziente HIV-positivo
- Memetica e nuovi media
- Peptidi overlapping derivanti da sequenze gH-omologhe di HSV-1 come inibitori dell'attività virale.
- Selezione di Mutanti HIV-1 Resistenti e Basi Molecolari della Resistenza di Nuovi Inibitori Non-Nucleosidici della Trascrittasi Inversa
Puoi scaricare gratuitamente questo appunto in versione integrale.